生命誌ジャーナル
2010年編む
4億年もRNAを書き換え続けてきた意味 由良 敬 お茶の水女子大学

1. 植物オルガネラの奇妙な現象から発見されたRNA編集
1980年代後半から、陸上植物の研究者は葉緑体やミトコンドリアなどの細胞小器官(オルガネラ)がもつ遺伝子発現の際に起きている妙なことに気がついていた。オルガネラのDNAとRNAの塩基配列に食い違いがあり、それぞれから推定できるタンパク質のアミノ酸配列が異なっているのである。最初は実験上の誤りだと思われていたが、研究の積み重ねにより、実は植物が積極的にRNAを編集していることが明らかになってきた。現在では、オルガネラDNA塩基配列から転写されたRNA分子に結合して特定の塩基を置換するタンパク質が存在し、その結果、成熟mRNAが生み出されることが分かっている。この過程をRNA編集と呼ぶ。つまり、オルガネラのDNA塩基配列をすべて解読しても、細胞内のRNA塩基配列とそれがコードするタンパク質の配列を知ることはできないのである。
このように、遺伝子が転写されるごとに書き換えるのは、間違いを起こしやすく手間がかかる危なっかしい仕組みに見える。オルガネラDNAに問題点があるのならば、DNAを修正すればすむことだが、陸上植物はなぜわざわざこんな面倒なことをしているのだろう。
2. 情報の変換の進化をRNA編集で捉える
私とRNA編集との出会いは1996年頃である。当時、名古屋大学におられた郷通子先生(現:情報・システム研究機構)のもとで、生物の情報の変換の進化の研究をすすめていた。たまたま、植物オルガネラDNAの専門家である杉浦昌弘先生の研究室を訪れていたRNA編集の専門家ハンス・ケッセル先生の話を聞き、これこそ情報変換の進化のテーマだと思ったのである。ケッセル先生は植物の光合成反応の中核となる酵素RuBisCO(註1)のRNA編集部位とそのRNAがつくるタンパク質の立体構造とに関係があるのではないかと考えていた。
その後の研究で、陸上植物のRNA編集では、主にRNA塩基配列のシトシン(C)がウラシル(U)に変わることや、RNA編集部位を認識するタンパク質が核のDNAにコードされていることが分かってきている。また、その編集部位についてのデータは莫大な量にふくれあがりつつあり、そこから、植物オルガネラのRNA編集の意味を見い出せるのではというところにきている。
註1:RuBisCO
二酸化炭素と水と光からブドウ糖をつくりだす光合成反応の中核となる、炭素を固定するための酵素のこと。地球上で最も存在量の多い酵素と言われている。
3. 立体構造を支える縁の下の力持ち
そこで、生成するタンパク質の立体構造に注目し、RNA編集によってDNA塩基配列からの推定とは異なるアミノ酸の種類を知り、そこでできるタンパク質の立体構造を視覚化できるデータベースRESOPS(註2)を構築した。
DNA塩基配列上でRNA編集が起きている場所と頻度とを調べると、そのほとんどがタンパク質をコードする領域で起きており、ひとつの遺伝子あたり平均約9.7箇所あることがわかった。RNA編集によって置換されるアミノ酸がタンパク質の活性部位に存在する例はほとんどなかったが、一方で、RNA編集が適切になされないと十分に機能しないタンパク質があることも実験で確かめられている。一体、RNA編集は何をしているのだろう。
データベースからRNA編集によってどんなアミノ酸に変化しているのかを調べたところ、923カ所のうち、セリンからロイシンが333回、プロリンからロイシンが325回、セリンからフェニルアラニンが248回と、この順に多いことが明らかになった(図1)。
註2:RESOPS
陸上植物のRNA編集データベース
http://cib.cf.ocha.ac.jp/
RNAEDITING/

図1:RNA編集によるアミノ酸の種類の変化
RNA編集の結果、最も多かったのはセリン(親水性アミノ酸)からロイシン(疎水性アミノ酸)の変化であった。アミノ酸の種類が変わらない場合もあれば、終止コドンがグルタミンやアルギニンに変わる例もあった。
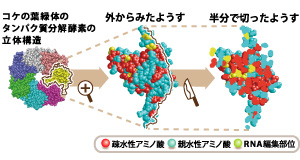
図2:タンパク質立体構造から分かるRNA編集の傾向
疎水性アミノ酸の多くはタンパク質中心付近に集まっている。
これは親水性アミノ酸から疎水性アミノ酸へと置き変わる傾向が強いことを示している。この変化がタンパク質の立体構造上どのような部位で起きているかをみると、置換されるアミノ酸は、タンパク質の中心付近に集まっている(図2)。一般に、疎水性アミノ酸は中心付近に局在するので、この事実は単に親水性から疎水性への置換の言い換えに過ぎない危険がある。
しかし、この効果をのぞいても、RNA編集によって置換されるアミノ酸は明らかにタンパク質の中心付近に集まっていた。編集されていないRNAが翻訳されると、タンパク質の中心付近に親水性アミノ酸が存在することになり、タンパク質の立体構造が不安定になる。立体構造を形成してはじめてタンパク質は機能するので、RNA編集はタンパク質の構造形成や安定性を通して機能を制御していると考えられる。
4. RNA編集をしなやかに利用した陸上植物
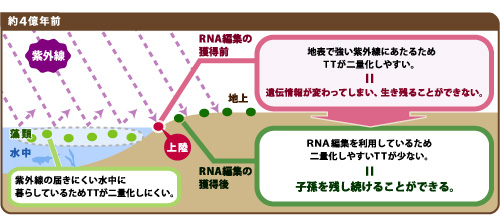
図3:植物上陸の際にRNA編集が果たした意味
二量化するTTを避けるような編集が多くなされることから、RNA編集は植物が上陸する際にオルガネラゲノムを紫外線から守る役目を果たしたとも考えられる。一方で、核ゲノムには水中の藻類にも陸上植物にもRNA編集が全くみられない。核ゲノムではDNA修復が効率よく行われているためであろう。
オルガネラゲノムにコードされているタンパク質の発現には、核にコードされているRNA編集用のタンパク質が必要不可欠らしい。つまり核がタンパク質の立体構造形成を通して、オルガネラの遺伝子の機能を調整しているのである。オルガネラの遺伝子の一部は核ゲノムに移行して発現調整を受けていることが知られているが、タンパク質の構造形成を通してのゲノムによる調整もあるということだ。RNAを編集するタンパク質が編集されるRNAがコードするアミノ酸配列を知ることはできないはずであり、編集部位の認識にはおもしろい機構が隠されているに違いない。大量のデータの中からその証拠を拾い出し、RNA編集の機構とその進化を明らかにしていきたい。
由良 敬 (ゆら・けい)
お茶の水女子大学大学院人間文化創成科学研究科教授。名古屋大学大学院理学研究科博士課程修了。名古屋大学理学部助手、日本原子力研究所計算科学推進センター研究員を経て2008年より現職。今回の成果は情報・システム研究機構の郷通子氏との長年の研究から生まれた。