年度別活動報告
年度別活動報告書:2012年度
脊索動物と節足動物の共通祖先を理解する 2−1 オオヒメグモの胚発生を支配する分子メカニズムの解明
小田広樹(主任研究員) 秋山-小田康子(特別研究員)
佐々木瑞希(奨励研究員) 野田彰子(研究補助員)
逸見なつき(大阪大学大学院生)
はじめに
多細胞動物は形態や発生様式に基づいて「門」と呼ばれる高次分類群(例えば、ヒトを含む脊索動物門や昆虫を含む節足動物門)に区分されているが、それぞれの分類群を特徴付ける形態が進化してきた過程やその進化の仕組みを理解することは簡単ではない。その大きな要因のひとつは、分類群を特徴づける、機能性の高い進化的に安定な形態であっても、その形態を形作るための発生過程やゲノム情報(遺伝子発現制御機構や遺伝子ネットワークなどを含む)は進化の過程で多様に変化しうることにある。このことは、もし祖先の状態から大きく変容してしまったゲノムを持つ動物種だけを対象として比較解析を進めた場合、生物進化の歴史を誤って再構成してしまう可能性があることを意味する。
例えば、体の軸に沿った反復構造の存在は、ヒトなどの脊椎動物を含む脊索動物門と昆虫やクモを含む節足動物門に共通して見られる特徴であるが、その反復パターンを形作る遺伝的仕組みに両者が同一起源であることを示唆するような共通性は見出されていない。しかし、だからと言ってその類似の形態的特徴が異なる起源から別々に進化したと断言することはできない。なぜなら、共通祖先において存在した反復パターンを形成する仕組みが、その機能的役割を保持したまま、進化の過程で遺伝子成分や遺伝子ネットワークの大きな変化を受けたかもしれないからである。このような可能性を考えると、それぞれの動物門の中での遺伝的仕組みの多様性の程度を大局的に把握することは、動物門間の関係を理解する上で重要である。また同時に、特定の形態を保持したまま発生システムがどのように進化しうるかを、システム生物学や数理生物学を取り入れて理解を深めることも中長期的に重要な研究テーマとなる。
このような観点に立って、これまで私たちは独自のモデル生物種として、節足動物門鋏角類オオヒメグモ (Parasteatoda tepidariorum, 旧名Achaearanea tepidariorum) を開拓し、節足動物の基本的体制(ボディープラン)に関わる発生メカニズムの解析を行ってきた。ショウジョウバエなどを含む既存のモデル生物からの知見と比較することによって、節足動物門の中での発生メカニズムの多様化原理を探究し、同時に、他の動物門との関係を考察してきた。オオヒメグモの新しいモデル生物としての有用性は、技術的利便性に加えて、この動物種によって得られる知見の意外性にある。昨年度までの研究で私たちは、ヘッジホッグシグナルとディーピーピーシグナルを介した細胞間のコミュニケーションが、クモのからだの軸を作るパターン形成に主要な役割を果たすことを明らかにした1)。とりわけ、ヘッジホッグシグナルに関する発見は、節足動物の多様化過程において発生メカニズムが大きな変化を受けたことを象徴的に示すものであった。オオヒメグモのヘッジホッグシグナルは胚全体の極性の形成を支配しているが、同様の役割はショウジョウバエでは転写因子のビコイドが果たしている2)。この「ヘッジホッグ vs ビコイド」の違いは、オオヒメグモ初期胚が細胞性であるのに対して、ショウジョウバエ初期胚が多核性であることとも整合性があり、両動物種間の初期胚パターン形成の比較解析は、形態の保守的進化の中で極端に異なる形態形成の仕組みがどのように進化できたのかを理解するための絶好の研究機会を提供する。加えて、ヘッジホッグシグナルに関連する遺伝子は多細胞動物に広く分布するため、このヘッジホッグシグナルを基軸とした比較解析の展開は、異なる動物門の体軸形成を互いに関連づけることを可能にするかもしれない。実際に、扁形動物門のプラナリアではヘッジホッグシグナルが再生時の頭尾軸極性の形成に関わっていることが示されていることは興味深い3)。
本年度私たちは、オオヒメグモ胚の研究を中長期的に発展させることを見据えて、技術開発に重点をおいて研究を進めた。一番大きな進展は、多色の蛍光色素による遺伝子発現解析が可能になったことである。それにより、同一サンプル内で複数の遺伝子の発現を同時に定量することが可能となった。さらに、細胞標識及びRNA干渉と、新しく成功した蛍光色素による発現解析を組み合わせることで、表現型の解析を細胞レベルで詳細に行うことができるようになった。これらの技術の向上は、遺伝子発現波のダイナミクスを制御する仕組みの解析や体軸形成の仕組みの解析に役立つ。また、次世代シーケンサーを用いたオオヒメグモゲノムの解析にも着手した。本年度の研究により、オオヒメグモの研究基盤が強化された。
結果と考察
(1)多重色蛍光in situハイブリダイゼーション法のクモ胚への適用と遺伝子発現の定量化
今年度私たちは従来のチラミド法を改良することにより、オオヒメグモ胚において複数種の遺伝子のmRNAを蛍光で同時に検出することに成功した。これまで私たちはアルカリフォスファターゼを用いた発色法によりmRNAを検出してきた。この方法は単色なら非常に安定した実験法であるが、3色以上の発色は難しいこと、2色で染色した際の細胞レベルでの分解能があまり良くないことや、定量性がないことなどの問題を抱えている。蛍光による検出は分解能や定量性の向上が期待できるが、これまでの私たちの試みでは検出可能なシグナルはほとんど得られていなかった。そこで、蛍光での充分な感度を得るためにin situハイブリダイゼーション法の各ステップの条件の見直しを行った。胚の固定時間とハイブリ液の組成を変更することと、過酸化水素水処理のステップを加えることで検出感度が向上することが分かった。特にハイブリ液にデキストランナトリウムを添加することで格段にシグナルが強くなった。さらに蛍光チラミドを実験室内で大量に合成し、チラミドの反応液もLauterらの論文4)を参考に改良することにより、充分な強度の蛍光シグナルを得ることができた。続いて複数種のmRNAを同時に検出するためにRNAプローブの標識に関して検討を行った。その結果、これまで使用してきたDIG標識とフルオレセイン標識に加えて、DNP標識とビオチン標識が使用できることが分かった。ただし、DNPはプローブ作製に多少の難があり、ビオチンは検出感度が他より落ちる。現在使用しているライカの共焦点レーザー顕微鏡の4本のレーザーの波長に合わせた蛍光チラミドが準備できたので、まだいくつかの問題点は残るが、最大で4種類の遺伝子の発現を同時に解析可能となった。また、3種類の遺伝子と核を検出する組み合わせや、2種類の遺伝子と核、ビオチン-デキストラン(細胞標識用)を検出する組み合わせも可能である。特に後者は胚性RNA干渉実験に非常に有効であると考えられる(図1)。
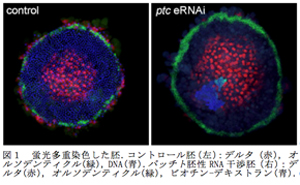
次に遺伝子発現の定量的解析を試みた。多重色蛍光染色した胚をライカの共焦点レーザー顕微鏡で観察して、三次元画像データを取得し、それらの画像データから画像解析ソフトのイマリスとImageJを使用して遺伝子発現の定量化をできるようにした。オオヒメグモ胚表面の細胞シートが曲面であることに対応するために、独自にJava(プログラミング言語)でImageJのプラグインを作成し、イマリスで指定した任意の線上の蛍光強度を数値として取得する方法を確立した。
(2)次世代シーケンサーによるオオヒメグモゲノムの解析
オオヒメグモをモデル種として効率的に研究を展開する上で、ゲノムワイドな遺伝情報の取得と解析は欠かせない。私たちは以前、サンガー法を基にしたシーケンシング手法でオオヒメグモ胚の転写産物の網羅的配列決定を行ったが、その網羅性は全く十分ではなく、発現量の低い遺伝子の配列を取得することが十分にできなかった。今回、次世代シーケンサー•ロッシュGS FLX+を用いることによって、より網羅性の高い配列情報が取得できることを期待して、再度オオヒメグモ胚の転写産物のシーケンシング解析を行った。できる限り多様な配列を取得するために、発生ステージの異なる6個の卵嚢から得たRNAを合わせてcDNA合成のテンプレートとし、ランダムプライマーを用いてcDNA合成を行った後、さらに均一化処理を行った。合計のリード数は890,221で、リードあたりの平均の配列長は433.49塩基であった。アセンブル解析で得られたコンティグ数は26,405であった。手持ちの低発現遺伝子の配列 (例えば、ソグやヘッジホッグなど) を用いて、これらのコンティグ配列に対してBLAST検索を行ったところ、今回のシーケンシングで取得した配列情報が低発現遺伝子の多くを網羅できていることが確認された。今後の研究にこのリソースを有効に活用していきたい。
転写産物のシーケンシングに加えて、オオヒメグモゲノムそのものも今年度新たに導入したイルミナ•次世代シーケンサーMiSeqを用いて独自にシーケンシングを行う準備を始めた。上述の転写産物の解析で用いた雌個体の別の卵嚢からゲノムDNAを精製し、300塩基前後に断片化してライブラリーを作製した。パイロット実験として25塩基x2のペアーエンドシーケンスを行ったところ、クオリティー値の高い約1ギガ塩基の配列情報が得られた。オオヒメグモのゲノムサイズは約1.4ギガ塩基であると推定されているので、250塩基x2のペアーエンドシーケンスを数回行えばゲノム全体を隈無くカバーするのに十分量の情報が得られることが期待される。
(3)頭部形成に関わるヘッジホッグ発現波の伝播を制御する分子メカニズムの解析
私たちは昨年度までの研究で、ステージ5において胚盤の縁に沿って1〜2列の細胞に出現するヘッジホッグの縞状の発現が、ステージ6の間にダイナミックに後方に向かって細胞から細胞へと伝播することを明らかにした。遺伝子発現波が伝播するこの現象は、オオヒメグモの頭部領域の形成に必須であり、ヘッジホッグと同じく胚盤の縁に発現するオルソデンティクルをRNA干渉によってノックダウンすると、ヘッジホッグの発現波の伝播が阻害され、頭部形成も妨げられる5)。
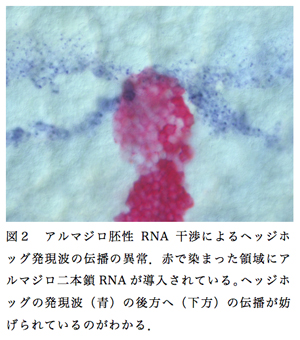
本年度、この現象を制御する分子的仕組みをさらに詳しく調べるために、様々な遺伝子に対して胚性RNA干渉を行うことによって、この現象に影響の現れる遺伝子を探索したところ、ショウジョウバエでアルマジロと呼ばれている遺伝子を見出した。アルマジロは脊椎動物のβカテニンに対応する分子で、カドヘリンと複合体を形成してアドへレンスジャンクションの構成因子として働く一方、ウイングレスシグナル経路において転写因子としても働く6)。今回オオヒメグモのアルマジロの胚性RNA干渉による実験では、ヘッジホッグのストライプが後方へ伝播してアルマジロの二本鎖RNAが導入された領域に接すると、ストライプはそれ以上後方へ伝播しなくなり、その後のストライプの分裂にも重大な影響が現れた(図2)。この表現型はアルマジロcDNAの、互いに重ならない3つの断片から作製した二本鎖RNAすべてで確認できたことから、アルマジロ遺伝子がRNA干渉によって特異的にノックダウンされた結果であることが強く示唆された。実際に、市販のヒトのβカテニンに対する抗体を用いた蛍光染色でも、アルマジロの二本鎖RNAの導入によりアドへレンスジャンクションに局在するアルマジロの発現が低下していることが確認された。しかし、アルマジロの胚性RNA干渉がよく効いた細胞クローン内であっても、かなりの量のアルマジロはアドヘレンスジャンクションに存在しており、Fアクチンの分布に影響が表れていないことから、観察されたヘッジホッグストライプパターンの異常はアドへレンスジャンクションの機能に関連する細胞運動の不調によるものというよりも、ウイングレスシグナル経路に関連する発現制御メカニズムの不調によるものであることが考えられた。
この考えと一致して、ウイングレスシグナル経路において負の制御因子として働くことが知られているアキシンの胚性RNA干渉でもヘッジホッグのストライプの挙動に影響が見られた。アルマジロの胚性RNA干渉の表現型とは反対に、後方に向かってヘッジホッグの発現が広がる傾向があった。以上のように、アルマジロとアキシンの胚性RNA干渉によって得られているデータは、頭部領域形成に関わるヘッジホッグ発現波の伝播にウイングレスシグナルが関わっていることを示唆した。現在、ヘッジホッグ発現波の伝播を制御する仕組みを理解するためにさらに詳細な解析を進めている。
(4)TALシステム活用の試み
TAL (Transcription activator-like) エフェクターは植物に対するバクテリア由来のタンパク質で、2つの可変アミノ酸を含む34アミノ酸からなるリピート構造を含み、1リピート対1塩基の対応関係でDNAの特定の塩基配列を認識することが知られている。その認識メカニズムを利用した改変タンパク質が、新しい遺伝子解析ツールとして注目されている。今年度はこのTALシステムを活用して、オオヒメグモで目的の遺伝子を異所的に発現させる実験系を開発することを試みた。
ヘッジホッグ遺伝子をオオヒメグモ胚で異所的に発現させることを目指し、まずヘッジホッグ遺伝子領域を含むゲノムDNAクローンを、ライブラリーをスクリーニングすることにより単離した。このクローンの全長約11kbの塩基配列を決定し、転写開始点の予測や遺伝的多型の有無の確認を行い、ヘッジホッグ遺伝子の発現調節領域として期待される配列を2つ選んだ。そして、これらの配列(それぞれを-50配列と-70配列と名付けた)を認識し、VP64の転写活性化ドメインをもつTALタンパク質をコードする遺伝子を作製し、in vitroでmRNAを合成した。現在、これらのmRNAをオオヒメグモ初期胚の割球のひとつに注入し、影響を観察しているところである。転写活性化型TALエフェクターは、標的とする配列や遺伝子による影響の違いが報告されている。さらに他の標的配列に対するエフェクターも合成し、その結果も合わせて、オオヒメグモに対する有効性を評価したいと考えている。
おわりに
本年度は技術開発に重点をおいて研究を進めた。多色の蛍光色素を用いて遺伝子発現解析が可能となり、定量的な解析を行う基盤ができた。このことは数理生物学やシステム生物学といった方向への研究の発展につながると期待している。また、次世代シーケンサーを用いたオオヒメグモゲノムの解析も開始した。これらの新たな取り組みを、ゲノム進化と形態進化の関係の探究に繋げていきたい。
