論文
Yasuko Akiyama-Oda, Takanori Akaiwa, Hiroki Oda
Reconstruction of the Global Polarity of an Early Spider Embryo by Single-Cell and Single-Nucleus Transcriptome Analysis
Front. Cell Dev. Biol. 10:933220. doi: 10.3389/fcell.2022.933220
オープンアクセス論文 (BB CY 4.0)
解説
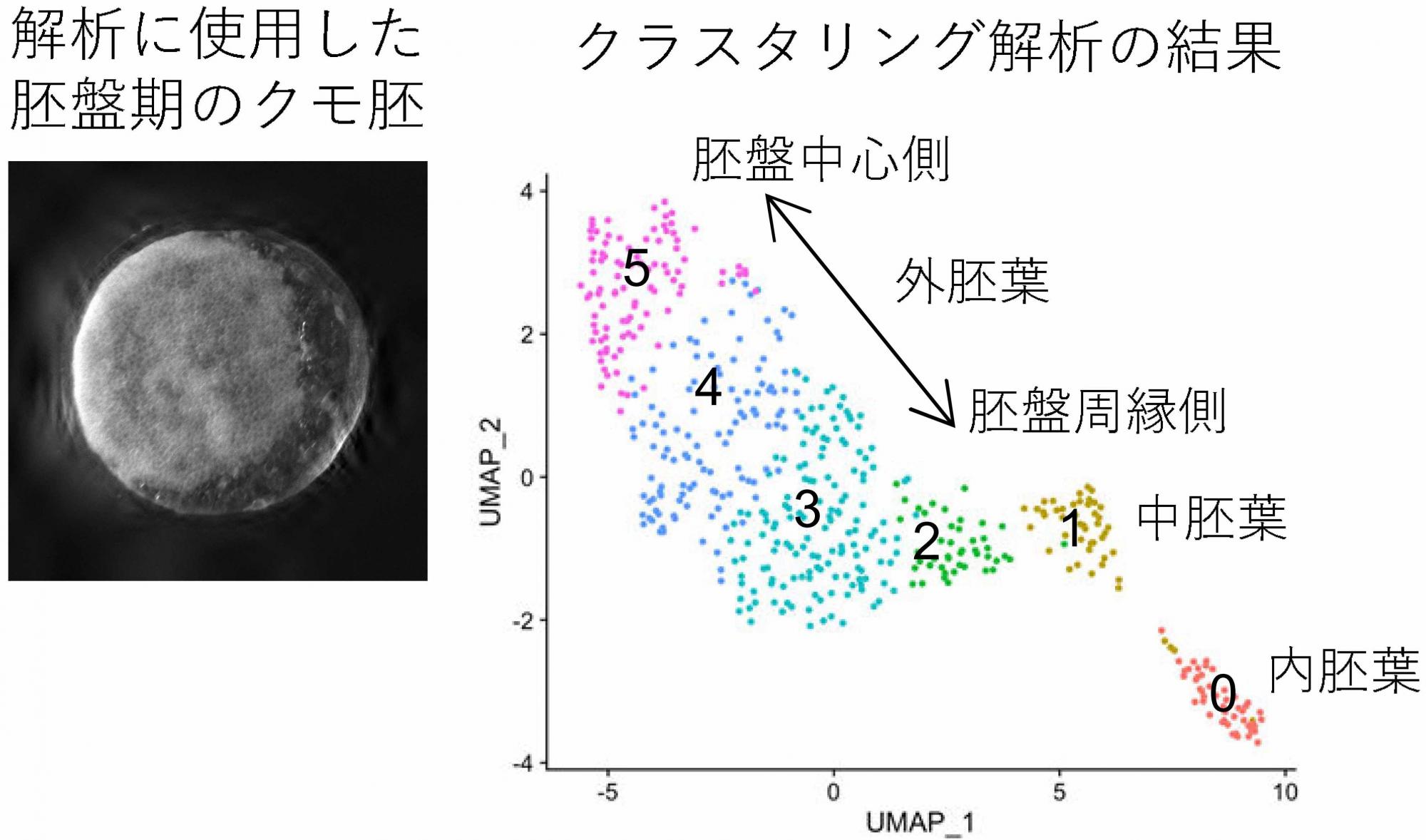
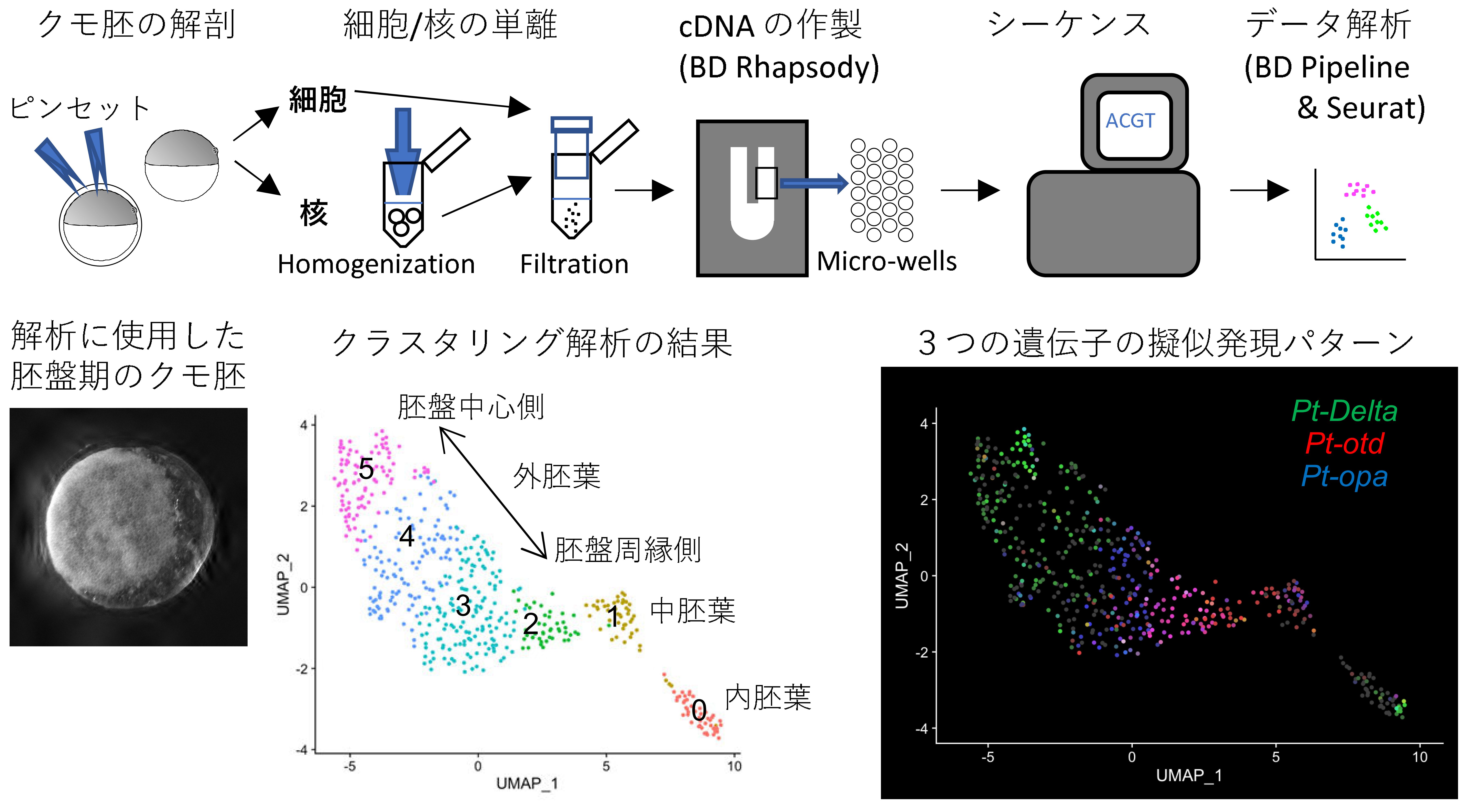
卵の中の細胞をバラバラにしても、からだの軸がコンピュータで再現できてしまう不思議
本研究では、シングルセルRNAシーケンシングとシングル核RNAシーケンシングと呼ばれる先進技術を胚盤期のオオヒメグモ胚に対して初めて適用し、これらの技術がクモ胚の中に生じる遺伝子発現のパターンを高い精度で再構成できることを明らかにしました。これまでの私たちの研究により、オオヒメグモ胚ではヘッジホッグシグナルと呼ばれる分子シグナル系がはたらいて、胚盤上に将来の体の前後軸に沿って遺伝子発現のパターンが形成され、これらの遺伝子発現をもとに将来の体の繰り返し構造が形作られることが分かっていました。しかし、これまでの研究では胚全体から抽出したRNAをもとに解析が行われており、1つ1つの細胞の状態を把握することはできていませんでした。今回用いたシングルセルRNAシーケンシングとシングル核RNAシーケンシングは、胚の細胞(または核)をバラバラにして個別化し、それぞれの細胞(または核)からゲノム上の全ての遺伝子に対して発現量の情報を取得する技術です(図上段)。両技術は一般的に、胚や組織の中の個々の細胞の状態を網羅的にかつ定量的に解析できる点で優れています。しかし、前処理で細胞をバラバラにしてしまうため、個々の細胞の位置情報を保持できないことに問題がありました。しかし、今回の私たちのクモ胚を用いた実験の結果では、胚表面の細胞層内に形成されつつある体の軸の方向が正しい順番で擬似空間にマップされました。つまり、取得した個々の細胞または核の遺伝子発現量の情報だけから元の細胞が存在していた位置関係がかなりの程度で再構成できることが明らかになりました(図下段)。この事実は、ゲノム上のすべての遺伝子の発現パターンを推定できることを意味します。また、類似の発現パターンをもつ遺伝子をコンピュータ上の検索で探し出すことも可能となります。今回の発見は、オオヒメグモの実験系が動物胚でのパターン形成の研究のために優れたモデルを提供しうることを示すもので、このモデル生物を用いた研究の進展が期待されます。
ラボニュースへ
