論文
Takanori Akaiwa, Hiroki Oda, Yasuko Akiyama-Oda
Genome-wide quantitative dissection of an arthropod segmented body plan at single-cell resolution
Communications Biology 8:913
解説
[最新論文] 節足動物のからだの基本構造をゲノムと数値データで解剖: クモから築く、多様性の起源を探究するための研究基盤
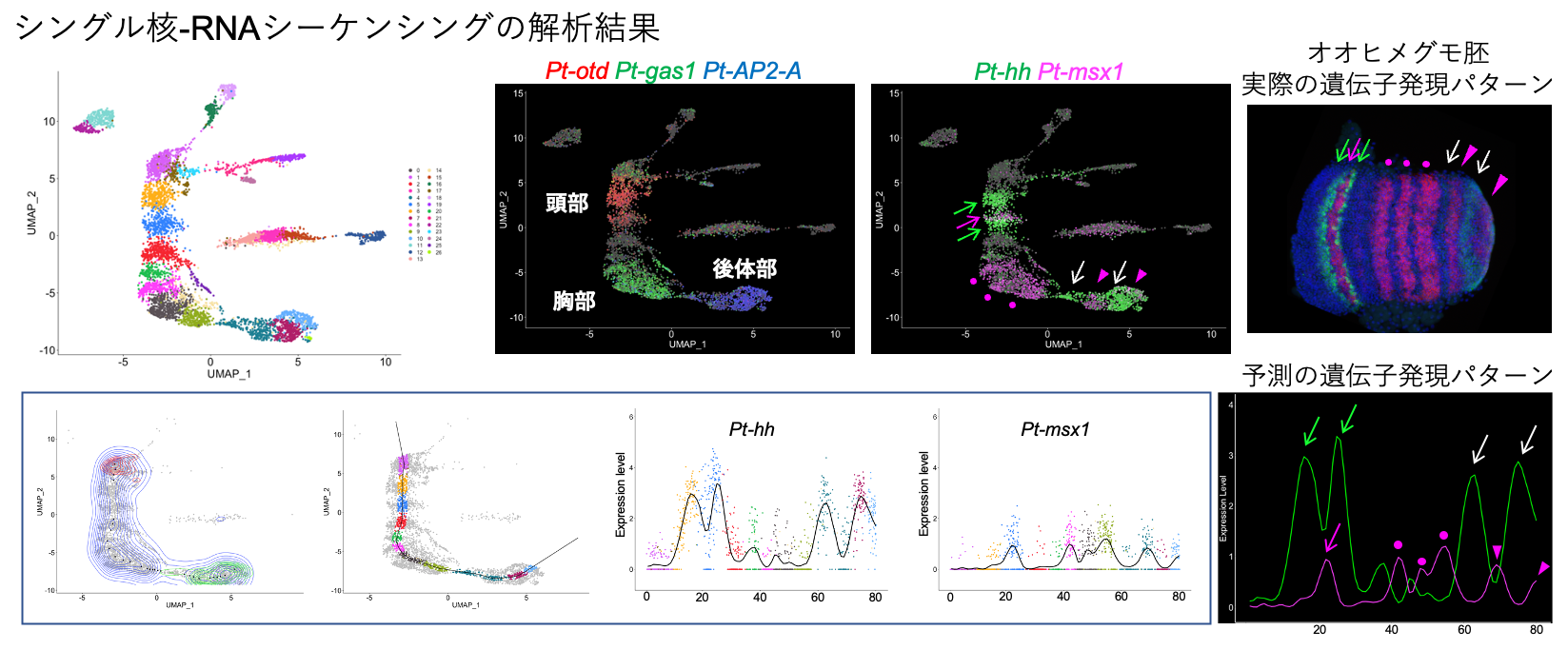
節足動物は共通してからだの軸に沿った分節構造を持ちます。しかし、その分節構造が形成される過程や仕組みは多岐にわたっています。節足動物の特徴を築いた、進化的に最初の仕組みはどのようなものだったのか、地球上における節足動物の多様性と繁栄を考えるとき、その問いは大きな意味を持ちます。多様な進化を可能とした起源となる動物の形づくりの仕組みを理解することは私たちの研究室の長期目標となっています。その目標に近づくための鍵は、ゲノムと、パターンや進化を客観的に扱うための数学です。今回の研究の成果は、ゲノムを基盤とした解析によって予備知識なしに節足動物の特徴を数値データで得られることを示しました。からだの形、ゲノム、数学を結びつけるための大きな一歩と考えています。本研究では、シングルセル/シングル核RNAシーケンシングと呼ばれる先進技術を用いて、体作りが進行するオオヒメグモ胚を構成する個々の細胞の状態を定量的に取得し、解析しました。
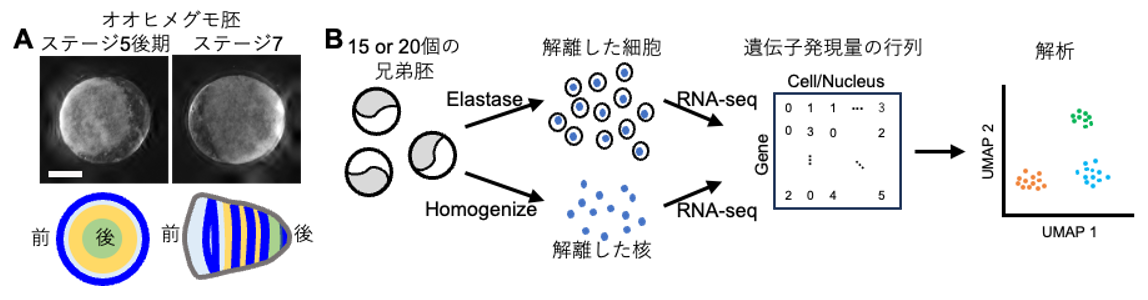
この実験では細胞の核をばらばらに取り出してから遺伝子発現量の取得を行うため、本来は遺伝子発現量と元々細胞が存在していた空間の情報を関連づけることはできないはずが、取得した個々の核の遺伝子発現量の情報だけから元の細胞が存在していた位置関係がかなりの程度で再構成できることが明らかになりました。クモ胚から取得した数値データのみの解析で、節足動物を特徴づける体の軸に沿った繰り返しパターンを再現しました。このことにより、ゲノム上のすべての遺伝子について体軸に沿った遺伝子発現のプロファイルを予測できることになりました。

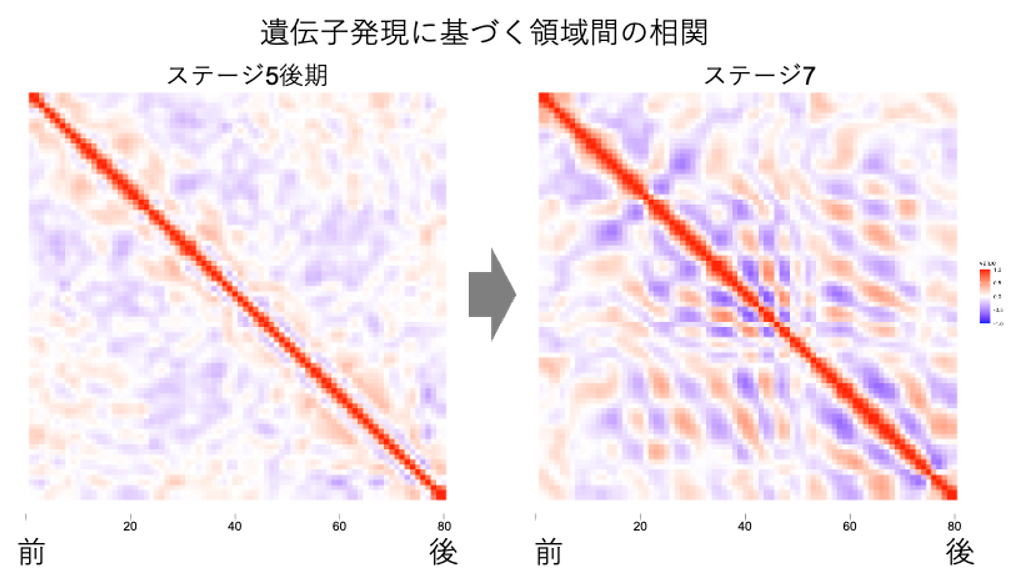
得られた数値を使って数学的、統計学的手法による様々な解析ができるようになります。例えば、空間の軸方向に沿って微分値を取れば、遺伝子の発現領域の境や遺伝子発現量のピークを捉えることができます。その微分値に基づいて異なる空間領域の間の相関を取ると似た領域の繰り返しが見えてきます。
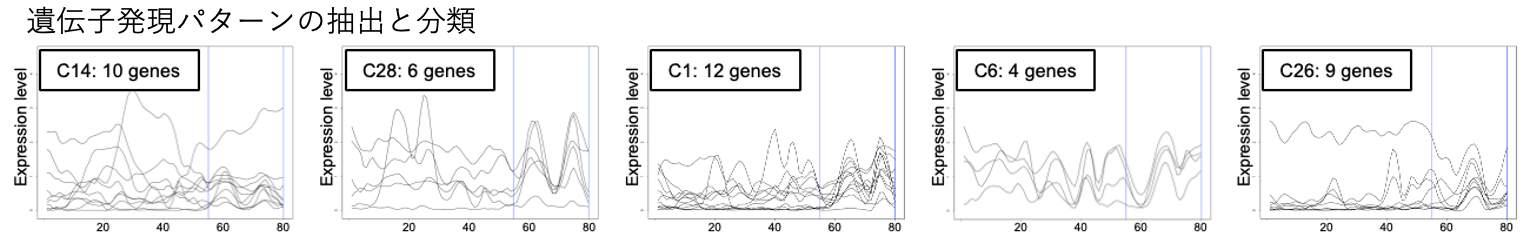
また、統計学の階層クラスタリングという手法を用いると類似の発現パターンをより分けて分類することもできます。
さらに、各細胞内の異なる遺伝子の間の発現レベルの相関を取ると、遺伝子と遺伝しの制御関係も見えてきます。下の図では円周上に遺伝子が配置されており、遺伝子間の相関を曲線で結んで示しています。赤色は正の相関で、青色は負の相関を示しています。
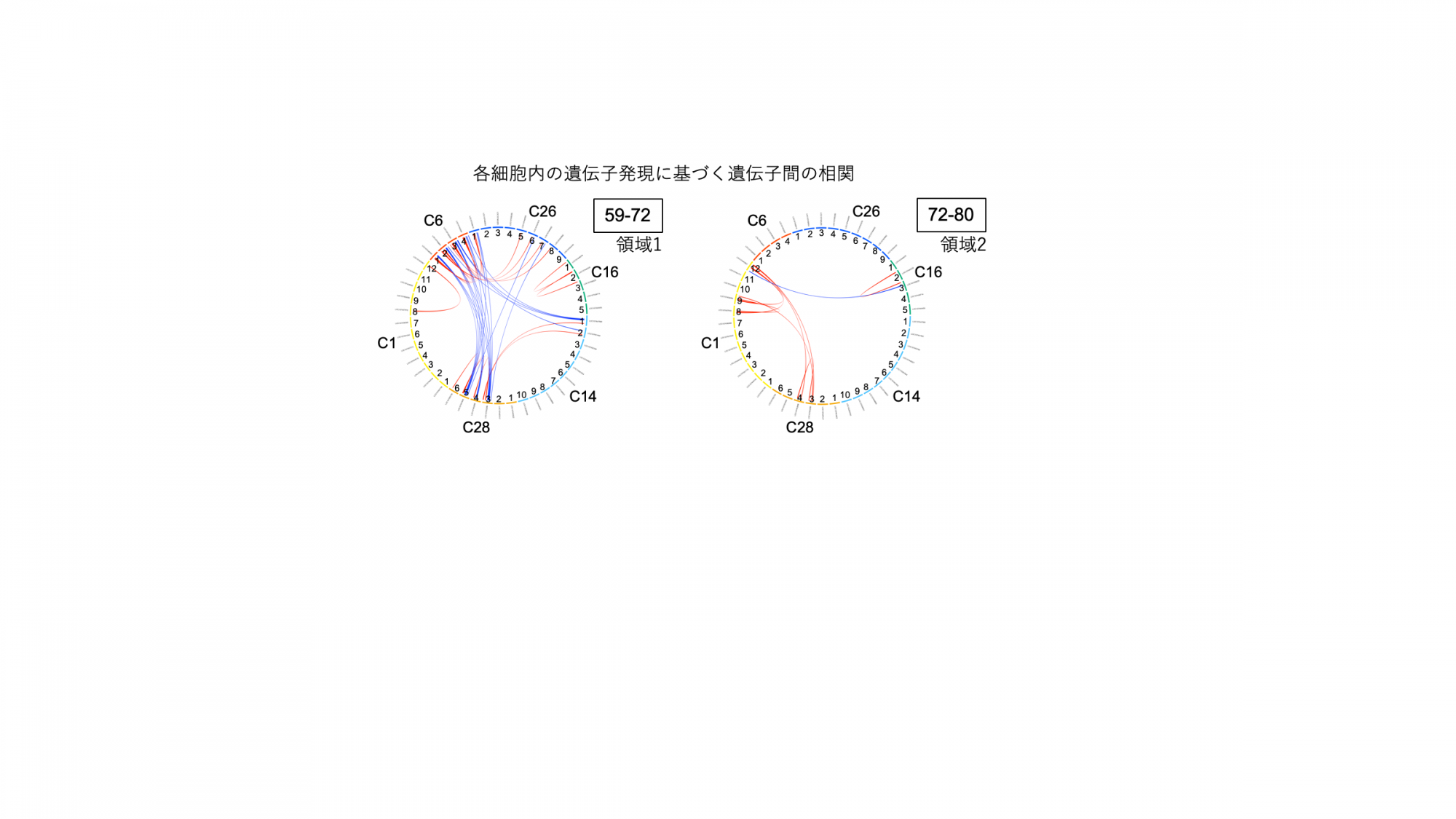
今回オオヒメグモ胚から取得した数値データは2つの発生段階だけからのものですが、たった20個のクモ胚を使うだけでこのようなデータが取れてきており、発生段階を細かく追ってデータを揃えることも可能となっています。実際に現在、そのようなデータの解析を行なっており、さらに発展した形で、節足動物の特徴を解析できる研究基盤が築けると考えています。データを共有する仕組みづくりにも取り組んでいます。こちらもご覧ください。
