年度別活動報告
年度別活動報告書:2003年度
アゲハチョウの食草選択と進化
吉川 寛(顧問)
小野 肇(奨励研究員)
尾崎克久(奨励研究員)
広崎由利恵(研究補助員)
はじめに
昆虫は寄主選択、配偶行動、集団生活の維持など様々な場面において化学物質を巧みに利用している。昆虫と環境との関わり、その結果として生じる多様化、種分化、進化の謎を解くために、嗅覚や味覚といった化学受容に関わる分子の解明が有力な手がかりになると考えられる。
アゲハチョウは単食性に近い食草選択をしており、食草選択とアゲハチョウ科の進化とに相関関係が認められる。このことに注目して、雌成虫は前脚ふ節に存在する感覚毛によって食草に含まれる化合物を識別して産卵することが明らかにされた。最も良く研究されているナミアゲハ(Papilio xuthus)では、ウンシュウミカンの葉から産卵刺激物質として10種類の化合物が単離されている1)。それらは、主としてフラボノイド、アミン類、糖の誘導体に分類される。ほかにも数種のアゲハ類で産卵刺激物質が明らかにされており、アゲハチョウの系統と産卵刺激物質の構造を比較すると、アゲハ類は特定の系統の化合物群を寄主認識の手がかりとしながら食性の幅を広げたと考えられる。感覚毛には典型的な味覚細胞が存在することから、味覚細胞には産卵刺激物質特異的な味覚レセプターが発現していることが予測される。アゲハチョウにおいて、味覚のレセプターは種ごとに特徴的な多様なレパートリーを持っていることが予想され、それらを網羅的に解析すれば、レセプター進化と食性の変化の関係を解き、種分化の謎に迫れると考えた。
昆虫の味覚レセプターの研究は2000年になって初めてショウジョウバエから候補遺伝子として新規の7回膜貫通Gタンパク質共役型レセプター(以下GPCRと略)ファミリーが同定された2)。以降、ハマダラカとオオタバコガについて化学感覚器官に特異的に発現するGPCRが発見されている3), 4)。これらのレセプターはその構造の多様さのため、クローニングは難しくいずれも全ゲノム配列情報からはじめて明らかにされたものである。また、嗅覚、味覚などの化学物質がリンパ液中の細胞表面に存在するレセプターに到達するために、これら化合物を結合し運搬する蛋白(Odorant Binding Protein=OBP)が知られている。これについても最近ショウジョウバエ全ゲノムの解析から昆虫には多種類のOBP遺伝子が存在し、味覚器官に特異的に発現するものも明らかになった5)。このように昆虫の味覚レセプターとOBPに関する配列情報が蓄積されつつあることは、情報科学的手法を用いてアゲハチョウの産卵刺激物質選択にかかわる遺伝子群をクローニングする道が開けた。一方昆虫には神経伝達に作用するアミン類のレセプターが知られている。面白いことにこれらのアミン類と同種、あるいは類縁体がアゲハの産卵刺激物質として作用している6)。味覚物質としてのアミン受容体と神経物質としての受容体は同種かどうかも興味深い課題である。又、02年度の研究で、ふ節特異的に発現するチトクロームp450が存在することを発見した。昆虫においてp450は摂食した植物成分を解毒する作用が知られている7)。ふ節で発現するp450は感覚毛に受容された産卵刺激物質を代謝分解する働きが予想されるので、解析の対象に加えた。
そこで、産卵刺激物質の全容が明らかにされているナミアゲハを材料に先ず前脚ふ節に発現する味覚レセプター遺伝子、OBP及びアミン受容体のクローニングを目的とした。初年度はアゲハ成虫前脚ふ節からのcDNAライブラリーの調製、大量配列方法の確立、RT-PCRによる発現部位決定法を確立し、2年度はcDNAクローンの網羅的な配列決定と、得られた配列の情報解析、発現解析を行った。その結果、雌ふ節に特異的なESTとして7回膜貫通型GPCRの部分配列(PXFT-0198)を、雌雄のふ節に特異的に発現す3種類のOBPと3種のアミンレセプター及び1種類のチトクロームp450の遺伝子をクローニングした。
今年度はcDNAライブラリーからの遺伝子発見は限界に達したと考え、ナミアゲハゲノムライブラリーを作製し、既に得られた上記候補遺伝子をプローブとして、ゲノム上の遺伝子の全構造と関連遺伝子ファミリーのクローニングを行った。また、これらの遺伝子群の機能解析を行うため、発現部位を細胞レベルで正確に決定するin situ hybridization 法を確立した。
結果と考察
1.アゲハ産卵刺激物質受容体候補PXFT-01989のゲノム配列
a)ショットガンシークエンス
Fosmidベクターを用い、ナミアゲハのゲノムライブラリーを作成した。約40Kbpのインサートを含む67200クローンを、384穴プレートで175枚フリーズストックとして保有している。ナミアゲハのゲノムサイズがカイコと同等の530Mbpであると仮定すると、全ゲノムの5倍の重複率となるクローン数である。
PXFT-01989の配列を含むクローンをゲノムライブラリーから検出し、遺伝子完全長配列の取得とホモログ遺伝子の探索を目的として、ショットガン法によりFosmidクローンの全長配列を決定した。塩化セシウム密度勾配超遠心法によりFosmidクローンを高純度に精製し、ソニケーションにより1~5Kbpの大きさになるよう断片化を行った。断片化したDNAを平滑化し、平滑末端を持つシークエンスベクターにライゲーションすることでショットガンライブラリーを作成した。LBアガープレートからコロニーを無作為に拾い、プラスミドDNAを精製し、クローニングサイト付近のプライマーを用い両方向から塩基配列を決定した。
塩基配列データからPhredを用いてベースコールを行い、アセンブルにはPhrap、Cap3、Gap4の3つのソフトウエアを用いた。これら3つのアセンブルソフトで解析結果が異なった場合、全てのケースでCap3の結果が正解であった。PhrapとCap3は両方向から配列を決定した場合、同一クローンのデータが向かい合ってContig上に配置するように考慮するため、アセンブルの精度が改善され全長配列を得るために必要なショットガンクローン数を抑えることができるという特徴がある。Cap3を用いて解析を行った場合、8倍の重複率で完全長、または1ギャップのデータを得られた。このことから、広範囲の塩基配列を効率よく決定することができたと考えている。
b)遺伝子構造とタンパク質高次構造の推定
Berkeley Drosophila Genome Project が提供している Splice Site Prediction アルゴリズム8)を用いエキソン領域を推定した結果、PXFT-01989の完全長と思われる配列が得られた。この遺伝子は、7つのイントロンによってコード領域が分断されていた(図1)。8つのエキソンを合わせた塩基配列の開始コドンから終止コドンまでの長さは1269bpとなり、422残基のアミノ酸配列が推定された。膜貫通領域を推定した結果、7回膜貫通型のタンパクであると予測された。BLASTによるホモロジー検索では、オオタバコガの化学物質GPCRである Chemoreceptor 4 に全長にわたって類似性を示した。この2遺伝子のコンセンサス配列によるPSI-BLASTでは、ショウジョウバエの味覚GPCRである Gr43a がヒットした。Supprt vector machines アルゴリズムを用いたGPCRの分類9)では、昆虫の化学物質受容体のサブファミリーであると推定された。
以上の結果より、PXFT-01989はナミアゲハの産卵誘導に関与するGPCRであると考えられる。ショットガン法により得られた周辺の配列から、GeneMark.hmm10)を用いて遺伝子領域を推定した結果、その他のGPCRのホモログを見つけることはできなかった。
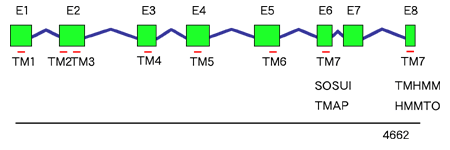 |
図1: PXFT-01989の遺伝子構造
イントロン(青線)により、エキソンが8つに分断されていた。エキソン部分(E1-E8)を集めると、開始コドンから終止コドンまでの長さが1269塩基、予想されるタンパクは422残基であった。4種類の膜貫通領域推定アルゴリズム(SOSUI・TMAP・TMHMM・HMMTOP)すべてで7回膜貫通タンパクであると推定された。7番目の膜貫通領域の場所のみは、 解析ソフトによって異なる位置に置かれている。TM1-6は膜貫通領域の位置(赤線)を示す。
2.化学物質結合タンパクのゲノム配列
a)ゲノム配列とゲノム上の配置
cDNAライブラリーから見つかった化学物質結合タンパク遺伝子は、システインが4つ保存されているOS-Dファミリーと、システインが6つ保存されているOBPファミリーの2種類あったが、ふ節に特異的に発現しているのは3種類のOS-Dファミリー遺伝子であった。そこで、OS-Dファミリーの遺伝子についてGPCRと同様に、ショットガン法を用いてゲノム配列を決定すると共に近傍に遺伝子ファミリーが存在する可能性を検討した。
40Kbのインサートについて、GeneMark.hmm10)を用いて遺伝子領域を推定した結果、cDNAライブラリーからは見つかっていなかった新規OS-Dファミリー遺伝子が5つ見つかった。推定されるアミノ酸配列には、先頭のシグナルペプチドと4カ所のシステインが保存されていた。ゲノム上で近くに存在し、同じ方向を向いている遺伝子同士でアミノ酸配列の類似性が高い傾向が観察された。現在、これら新規に発見した遺伝子群の発現部位を検討している。
|
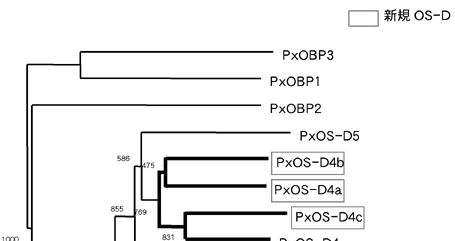
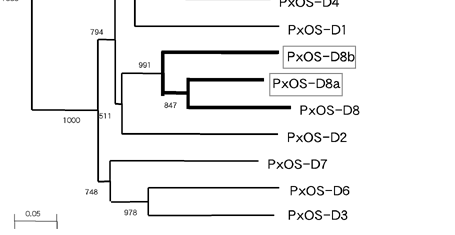
図2:OS-D・OBP遺伝子のクラスター解析
EST解析で見つかっていた既知OS-D・OBPと、ゲノム解析で見つかった新規OS-Dのアミノ酸配列を用い、クラスター解析を行った。新規に見つかったOS-D遺伝子は、ゲノム上で近くに存在し同じ方向を向いていた既知遺伝子と単系統群となった。1000回反復のブーツストラップ値は、おおむね高い値を示している。OS-DファミリーとOBPファミリーは最初に分岐し、独自に進化したことを示している。
b)OS-Dファミリー遺伝子の分子系統
新規OS-D遺伝子と既知のOS-D・OBP遺伝子を用い、近隣結合法によるクラスター解析を行った結果、ゲノム上で近くに存在し同じ方向を向いている遺伝子が単系統群を形成した(図2)。この結果から、OS-D遺伝子はゲノム上で重複し、類似する機能を持つファミリー遺伝子がクラスターを形成しているのではないかと考えられる。
3.アミンレセプターの発現部位の解析
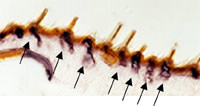
産卵刺激物質には神経伝達物質のアミン類が含まれることから、産卵刺激物質のレセプターの中でも、アミン類に対するレセプターは神経伝達に働くレセプターと共通性を持っているかも知れない。このような可能性を検討する目的で、アゲハにおいて発現する神経伝達物質(アミン)レセプターの解析を行った。昨年度までに、セロトニン、オクトパミンおよびドーパミンのレセプターのクローニングについて報告した。本年度は、ふ節の凍結切片作製とin situ hybridizationの技術を確立して発現部位について解析した。その結果、ドーパミンレセプターについて感覚毛の味覚細胞に相当する部位での局在的な発現が認められた(図3)。これよりドーパミンレセプターが産卵刺激物質の受容に関与していると考えられる。化学受容の際にドーパミンレセプターが果たす役割として次の二つの可能性が考えられる。1)直接、産卵刺激物質を受容する。2)シナプス部で神経伝達物質を受容して産卵刺激に対する興奮あるいは抑制に関与する。これらの可能性を検討するためには、タンパク質の発現解析によるリガンドの同定、電気生理学的手法を用いた神経伝達物質に対する味覚細胞の応答の解析、といった手法を用いる必要がある。
|
Anti-sense probe
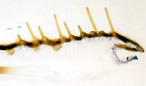 |
Sense probe (control)
図3:in situ hybridizationによるドーパミンレセプターの発現部位の解析
4.アゲハの化学感覚器に特異的に発現するP450遺伝子のクローニングとその発現部位の解析
 |
 |
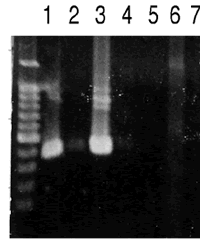
図4:RT-PCRによるP450の発現部位の解析![]()
EST解析の結果、3種類のP450がふ節で発現していることを確認した。植食性昆虫ではP450は植物二次代謝物質の分解・解毒に働いていることから、ふ節感覚毛で発現しているP450は化学受容後の産卵刺激物質を分解しているのではないかと考えて、発現部位の解析を行った。 RT-PCRにより3種類の遺伝子の各組織における発現の有無を解析した結果、1つのP450についてふ節と触覚における特異的な発現が認められた(図4)。この遺伝子の全長配列を決定した結果、既知のショウジョウバエ、ハマダラカやヨトウガ11)のCYP4ファミリーに属するP450とアミノ酸配列で35%程度の相同性を示した(図5)。
|
|||||
| 図5:アゲハ感覚毛特異的P450と他の昆虫のP450とのアミノ酸配列比較 |
さらに、ふ節の凍結切片についてin situ hybridizationにより発現パターンを調べた結果、この遺伝子は感覚毛の味細胞に相当する部位に局在して発現していた(図6)。以上の結果より、産卵刺激物質や匂い物質が化学感覚器で受容された後の脱感作の過程で,リガンドの分解にP450が作用すると考えられる。現在、大腸菌を用いたタンパク質の発現を行っている。今後、このP450について産卵刺激物質に対する酵素活性の解析を行う予定である。
|
Anti-sense probe
 |
| Sense probe (control) |
| 図6:in situ hybridizationによるP450の発現部位の解析 |
おわりに
ナミアゲハ雌前脚ふ節に特異的に発現する受容体様のタンパクは、ゲノム遺伝子の全体構造の解明によって、7回膜貫通型のGPCRで一次構造、高次構造、から化学物質受容体の一種で、既知の受容体の中ではショウジョウバエの味覚遺伝子の一種に類似することが明らかになった。これらの結果はこの受容体がこれまで知られていなかったアゲハの産卵刺激物質受容体の有力な候補であるといえる。この結果をより確実にするには、発現部位を細胞レベルで特定すること及び受容体のリガンドとなる化学物質を同定する必要がある。前者については、感覚毛領域のふ節の切片を用いてアミンレセプターとp450の発現を細胞レベルで同定するin situ hybridization 法を開発することに成功した。現在発現が少ないことが予想される味覚受容体の発現を測定するより高感度の方法の改良を工夫している。残念ながらこの受容体が存在するゲノム領域には約50Kbpの範囲にGPCR遺伝子を発見することが出来なかった。したがってこの受容体遺伝子が遺伝子ファミリーを作っているかどうか、今後に残された課題である。一方化学物質結合タンパクについてはふ節に特異的に発現する2種類の遺伝子にはそれぞれ近傍のゲノム領域に類似の構造を持つ遺伝子を複数ファミリーとして存在することが明らかになった。これらの遺伝子は共通の機能をもつことが予想されるので、それらの発現について解析中である。
アミン類の受容体は構造からみる限り、神経伝達に働いている内在性のアミン類の受容体と相同であると考えられる。これらがふ節にも発現していることは、同じ受容体が異所発現することによって、産卵誘導受容体として異なる機能を担っている可能性が考えられる。リガンドの決定など機能解析によって実証しなければならない。チトクロームp450についても、精製タンパクによるin vitro機能解析によって味覚細胞における機能の推定を行う必要がある。
このように産卵物質認識の研究はそれにかかわるタンパク群をESTおよびゲノム遺伝子解析によって網羅的にクローニングし、情報科学的な解析から構造と機能を研究する方法を継続すると共に、タンパクの機能を検証する方法を開発する方向に向かう必要がある。In situ hybridization については見通しがえられたので、高感度をめざして改良をはかる。また、受容体と化学物質結合タンパクの機能解析については、動物、昆虫細胞を用いたin vitro発現系の構築、RNAi などによる機能阻害実験などを試みる。一方、すでに発見した遺伝子群を近縁のアゲハチョウゲノム遺伝子と比較することによって、遺伝子の変化を知ると共に構造の保存状況からタンパクとゲノムの発現制御領域の重要な機能領域を推定することが可能である。比較ゲノム的な研究を展開したい。
参考文献
1 Nishida R, Ohsugi T, Kokubo S, and Fukami H. (1987) Oviposition stimulants of a Citrus-feedingswallowtail butterfly, Papilio xuthus L. Experientia 43:342-344.
2 Clyne PJ, Warr CG, Carlson JR. (2000) Candidate taste receptors in Drosophila. Science. 287:1830-1834.
3 Hill CA, Fox AN, Pitts RJ, Kent LB, Tan PL, Chrystal MA, Cravchik A, Collins FH, Robertson HM, Zwiebel LJ. (2002) G protein-coupled receptors in Anopheles gambiae. Science. 298:176-178.
4 Krieger J, Raming K, Dewer YM, Bette S, Conzelmann S, Breer H. (2002) A divergent gene family encoding candidate olfactory receptors of the moth Heliothis virescens. Eur J Neurosci. 16:619-628.
5 Galindo K, Smith DP. (2001) A large family of divergent Drosophila odorant-binding proteins expressed in gustatory and olfactory sensilla. Genetics. 159:1059-1072.
6 Nishida R, Ohsugi T, Fukami H. (1990) Oviposition stimulant activity of tryptamine analogs on a Rutaceae-feeding swallowtail butterfly, Papilio xuthus. Agric. Biol. Chem. 54: 1853-1855.
7 Feyereisen R. (1999) Insect P450 enzymes. Annu. Rev. Entomol. 44:507-533.
8 Reese MG, Eeckman, FH, Kulp, D, Haussler, D, (1997) Improved Splice Site Detection in Genie. J Comp Biol 4(3), 311-23.
9 Karchin R, Karplus K, Haussler D. (2002) Classifying G-Protein Coupled Receptors with Support Vector Machines, Bioinformatics 18(1):147-159.
10 Lukashin A.V, M. Borodovsky (1998) GeneMark.hmm: new solutions for gene finding. Nucleic Acids Research, 26, 1107-1115.
11 M![]() b
b![]() he-Coisne M, Jacquin-Joly E, Fran
he-Coisne M, Jacquin-Joly E, Fran![]() is MC, Nagnan-Le Meillour E. (2002) cDNA cloning of biotransformation enzymes belonging to the cytochrome P450 family in the antennae of the noctuid moth Mamestra brassciae.. Insect Mol. Biol. 11:273-281.
is MC, Nagnan-Le Meillour E. (2002) cDNA cloning of biotransformation enzymes belonging to the cytochrome P450 family in the antennae of the noctuid moth Mamestra brassciae.. Insect Mol. Biol. 11:273-281.