顧問の西川伸一を中心に館員が、今進化研究がどのようにおこなわれているかを紹介していきます。進化研究とは何をすることなのか? 歴史的背景も含めお話しします。
バックナンバー
次世代シークエンサー
2014年8月1日

今回は約束通り次世代シークエンサーの話だ。今現在使われているのに次世代シークエンサーとは少し変だ。おそらく普及や利用の進展が早すぎて、名前を付け替えるのが間に合わなかったようだ。やはり何となくおかしいので、現在はハイスループットシークエンサーと呼ばれることが多い。名前はどうであれ、この機械は生命科学にとどまらず世界を変えている。
言うまでもなく私たちの身体で働いている蛋白や機能的RNAの情報はDNA上の塩基配列としてコードされている。これが読めると、細胞や個体で発現しているタンパク質やRNAを情報として表現、蓄積、利用することが可能になる。ただこの配列を読む方法が開発されたのは1975年ちょうど40年前のことだ(図1)。

図1サンガー法によるDNA配列決定法を述べた最初のJ.Molecular Biologyの論文。右上の写真はサンガー博士。
既にアミノ酸配列決定でノーベル賞に輝いたサンガー博士が開発した、chain termination法(サンガー法:註)だ。その後、1977年アメリカアカデミー紀要にこのサンガー法とともに、もう一つのマキサム・ギルバート法(化学分解を用いる)が発表される。この業績に対して1980年、サンガーにとっては2度目のノーベル化学賞がギルバートとともに与えられている。実際私の世代でDNA配列決定と言うと、どちらかの方法を使った。私はサンガー法を使っていたが、最初はキットなど存在せず、deoxy対dideoxy核酸の最適比を決める時、レントゲンフィルムの上にラダーと呼んでいた配列パターンが見えるかどうかいつもドキドキしたのを覚えている。その後、アイソトープは使っていたが、条件を振らなくともキットを使えば誰でも比較的簡単にDNA配列が読めるようになった。図2は熊本大学で大理石病遺伝子を見つけた時(Nature345,442,1990)のゲルだが、Tが一本多くなって正常なM-CSF蛋白が出来なくなっているのが一目瞭然でわかる。このパターンを見つけたときは教室全員で興奮したのを覚えている。
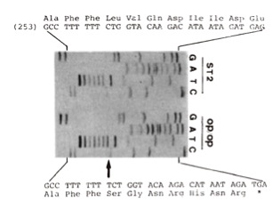
図2大理石病マウス突然変異を発見した時のシークエンスパターン。矢印はTの挿入を示している。
熊本大学から京都大学の分子遺伝学に移った頃(1993年頃)には既に蛍光プローブと毛細管に充填したゲルを用いた自動シークエンサーが普及していた(図3)。
レントゲンフィルムの上に現れたラダーと格闘しなくても、パソコンから配列がそのまま出てくるのをみて大喜びだった。この全自動シークエンサーが開発されて、様々な動物の全ゲノム解読が現実のこととなった。お金と時間さえあれば、シークエンサーを並べてヒトゲノム30億塩基対の塩基配列を解読できる。アメリカ合衆国は1990年から15年、30億ドルのプロジェクトとしてヒトゲノム計画を始めた。いつこのプロジェクトが完成したかを明確に示すのは難しいが、2001年にはおおよそのドラフト配列が、そして2004年に全ゲノムの99%以上を0.001%のエラー率でカバーしたと報告している。

図3 上:蛍光プローブを使った自動シークエンサー。原理はサンガー法と同じで、レントゲンフィルム上のラダー(左図)の代わりに、蛍光ラベルされた核酸が自動的に読まれている(右図)。下:両方の機器の写真。Wikimedia Commonsより。
さて、このプロジェクト進行中から、全く新しい様々な配列決定機器の開発が進められていた。様々な方法が開発されたが、これまでの方法とは次の2点で異なっていた。
- 1)電気泳動を使わず、配列を決めたいテンプレートに対応して伸長する核酸断片にA,T,C,Gそれぞれのどれが結合するかを直接蛍光観察する、
- 2)マイクロカプセルやプレート上で、何十万、何百万ものDNAの配列決定を同時に行う。
この結果一回の運転で、一度に何十万種類ものDNA断片の配列を決められるようになった。現在の最新鋭機を使うと、昔の機器なら何年もかかったヒトゲノムの解読を1日で終えられるようになっている(図4)。このおかげで図4右に示すように、2008年から配列決定にかかる費用は急速に低下し、ヒト全ゲノムの解読に要する費用は100万円を切った。また詳しく紹介する余裕はないが、これまでとは違う新しい原理を使って配列決定にかかるコストを劇的に下げようと言う試みも完成しつつあり、今やヒトゲノムの全配列を読むだけなら10万円を切るのも時間の問題になりつつある。おそらく10年も経てば、ヒトゲノムを調べるコストは1万円近くになるのではないだろうか。
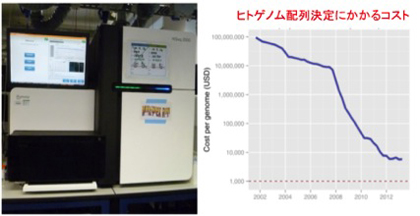
図4 次世代シークエンサーの一つ、イルミナ社のHiSeq(左図)。右図はヒトゲノム解読コストを年次別にプロットしている。1万ドルあたりで足踏みが始まっているが、新しいシークエンサー技術が開発されており、更なるコストの低下が期待されている。Wikimedia Commonsより。
このように核酸配列を読むためのコストが急速に下がったおかげで、興味のある動物のゲノム配列を気軽に行うことが出来るようになった。この結果、全ゲノム配列が明らかにされた生物は既に200を超えている。次回からは、次世代シークエンサーを駆使して行われた進化研究について紹介する。
註:サンガー法
目的の1本鎖DNAを鋳型としてDNAを合成する際、基質となるdNTP(デオキシヌクレオシド三リン酸)に対し、一定の割合で反応を止まるddNTP(ダイデオキシヌクレオチド)を混ぜて様々な長さの断片をつくる。この反応をA、T、C、Gそれぞれに対応するddNTPを使って行い、1塩基の差で分けられる電気泳動装置で分離しDNA配列を明らかにする手法。
![[科学のコンサートホール]BRH JT生命誌研究館](/common/img/logo.svg)